染色质免疫共沉淀(ChIP)技术
染色质免疫共沉淀(ChIP)技术
第一部分,什么是ChIP技术
1.1简介
染色质免疫共沉淀技术(Chromatin Immunoprecipitation, ChIP)是一种检测在体内自然染色质环境下蛋白和DNA相互作用的有效方法。这种技术广泛应用于检测特定基因调节蛋白结合在基因组中的具体位置或者基因调节区域和蛋白的修饰是否相关。。因其能真实、完整地反映结合在DNA序列上的靶蛋白的调控信息,是目前基于全基因组水平研究DNA-蛋白质相互作用的标准实验技术,日益成为研究真核细胞中转录调控情况的重要途径。
1.2技术原理
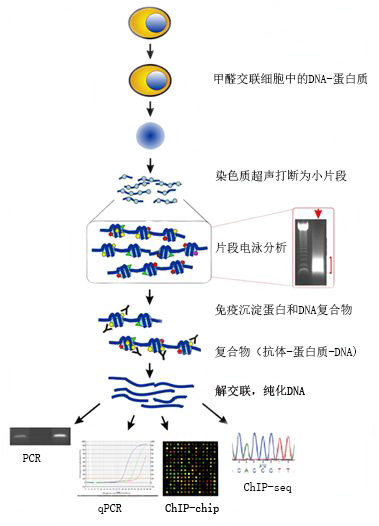
在活细胞状态下,通过在特定时间点上用甲醛交联等方式固定蛋白质-DNA 复合物,相当于这一时间点上细胞内蛋白和DNA相互作用的关系被瞬时“快照(snapshot)”下来。并将其用Bioruptor超声波破碎仪随机超声打断为一定长度的染色质小片段,然后通过免疫学方法将蛋白质-DNA复合物与特定DNA结合蛋白的抗体孵育,将与抗体特异结合的蛋白-DNA复合物洗脱下来,最后将洗脱得到的特异DNA与蛋白解交联,得到特异性地富集目的蛋白结合的 DNA 片段,,,通过对DNA片断的纯化与检测,从而获得蛋白质与 DNA 相互作用的信息。
1.3适用研究
1.31 筛选调控蛋白在基因组上的结合靶点:针对转录因子、DNA/RNA 聚合酶、转录复合物等调控因子蛋白进行特异性的富集,分离纯化与之结合的靶 DNA 片段并测序,分析筛选到准确有效的靶位点、靶基因数据;
1.32 揭示差异化表观遗传变异的原理:通过对不同表型组织内组蛋白、转录因子蛋白及其他结合蛋白进行染色质免疫共沉淀测序并定量分析靶基因表达调控水平或针对同种组蛋白进行甲基化、磷酸化、泛素化修饰所导致的结合位点变异进行分析,即可准确揭示差异化表观变异原理;
1.33研究表观遗传变异疾病:借助染色质免疫共沉淀测序技术结合生物信息分析揭示由蛋白与 DNA 相互作用变异而引起的疾病的原理,明确表观遗传修饰在癌症等表观遗传变异疾病的发生发展过程中的具体作用机制。
第二部分,ChIP实验具体如何操作
ChIP实验可用于研究植物动物微生物,对于动物样品有的是组织块(比如肿瘤样品),有的是黏附细胞或者悬浮细胞,根据具体实验对象ChIP实验的步骤流程和复杂程度会有些不同,大致上是相同的。我们可以用最常用的相对简单的动物黏附细胞作为实验对象阐述一下具体实验方法。
2.1甲醛交联细胞
(1)细胞在100mm培养皿上生长到80% ~90%。当第一次使用细胞系做实验时,另外培养两个培养皿,一个用于预测细胞数量,另外一个用于优化超声条件。
(2)去除上清培养液,加入lOmL 1xPBS含有终浓度为1%甲醛(加入270μL 37%甲醛到10 mL PBS)固定交联细胞,轻柔混合后置于室温10 ~20 min。
(3)加入1 mL 1.25mol/L甘氨酸(终浓度为0.125 mol/L)终止交联反应,甘氨酸终止甲醛的固定作用是通过提供过量的氨基团与甲醛结合。继续放在室温5 min。
注意:从这一步开始,把所有东西放在冰上或者4℃,包括离心步骤。
(4)吸取走上清液后,用10 mL预冷1xPBS洗两遍后,以去除残留的甲醛。
(5)加入4 ~5mL预冷1xPBS,能足够覆盖培养皿的表面,再用细胞刮棒刮落细胞, 转移到50 ml锥形离心管,再用4~5 mL预冷1 xPBS冲洗培养皿以收集残留的细胞。
(6)4000 rpm 离心 5 min (4℃)。
(7)去除上清,用1 mL预冷1xPBS (新鲜加入1xPIC 1O μL)悬浮细胞后,转移到1.5 mL离心管。
(8)4000 rpm 离心5 min (4℃)得到细胞沉淀,去除上清(细胞沉淀可放于 -80 ℃冰箱保存)。
2.2染色质超声断裂
(1) 加入1 mLSDS裂解溶液重悬沉淀(新鲜加入1 X PIC 1O μl,在冰上放置10 min,每隔1 min颠倒摇动离心管。SDS能裂解细胞膜和核膜,使固定染色质释放出来, 这样有利于超声。
(2) 用公认的标准化仪器Bioruptor超声仪把DNA打碎为长度在200 ~ 1000 bp,把样品放在冰上操作。不同细胞样本都得进行超声条件优化实验。
(3) 在超声后,14000 rpm 离心 10 min(4℃),转移超声染色质液体上清到1.5 ml 离心管。上清液可放于-80℃冰箱保存。
2.3免疫沉淀蛋白和DNA复合物
(1)使用ChIP稀释溶液把超声染色质液体稀释10倍,这样有利于免疫沉淀反应。 例如200 μL超声染色质液体加入1800 μL ChIP稀释溶液(新鲜加入1 x PIC 20 μL)。
(2)为了减少非特异性结合蛋白背景,往2 mL超声稀释液中加入20 μL蛋白A/G琼脂糖珠(Protein A/G Agarose Beads),在4℃摇床轻柔摇动1 h。
(3)1000 rpm离心2 min (4℃),转移上清液到新的离心管,加入1〜4叫免疫沉淀抗体(每种抗体加入的量都不同)到上清液中,在4℃摇床轻柔摇动过夜。另外取 50 μL上清液,不加入抗体,作为阴性对照,以验证检测的特性。
2.4收获免疫复合物(抗体-蛋白质-DNA)
(1) 40 μL Protein A/G Agarose Beads 先用 1 mL 1 x PBS 洗 3 遍,每次洗后 1 000 rpm 离心1 min,小心吸除上清,注意不要吸走Beads。最后加40 μL 1XPBS悬浮Beads。
(2)打开样品的管盖,加入40 μL Protein A/G Agarose Beads。在摇床上面摇动1~2 h,4℃,以形成抗体-蛋白A免疫复合物。
(3)1000 rpm离心5min,4℃。去除上清,注意不要吸走Beads。
(4)清洗Beads:免疫沉淀复合物依次用以下所列的缓冲液洗(每个1 mL)。每次洗时,使样品在摇床上面10min,4℃,然后再4000 rpm离心5 min。吸取上清时要注意不要吸走beads。
a.低盐洗脱溶液1 mL洗一次;
b.高盐洗脱溶液1 mL洗一次;
c.氯化锂洗脱溶液1 mL洗一次;
d.TE溶液(pH 8.0) 1 mL洗两次。
2.5洗脱免疫复合物
注意:从这一步开始,所有操作都在室温进行。
(1)加入200 μL ChIP洗脱溶液洗脱Beads,放在摇床上室温摇动10 min。12000 rpm,离心1 min,把上清转移到新的1.5 mL离心管。
(2)再加入200 μL ChIP洗脱溶液洗脱Beads,放在摇床上室温摇动10 min,一共洗脱3次,得到600 μL洗脱液。洗脱液中含有目的蛋白以及其结合的相关DNA。
2.6去除甲醛交联
(1)加入 24μL 5mol/L NaCl 到 600μL 样品中(NaCl 终浓度为 0.2mol/L),包括 上述1.3中的50 μL阴性对照(加入350μL ChIP洗脱溶液和16 μL 5mol/L NaCl)。
(2)65℃,过夜去除甲醛交联。甲醛能够在有盐离子和去污剂的高温环境下解除交联反应,使得蛋白质和DNA分离。
2.7 DNA 纯化
DNA纯化可以通过酚/氯仿抽提或者通过硅胶柱纯化。
酚/氯仿抽提
(1)对所有实验样本(600 μL)和50μL 阴性对照样本,分别加入 10μg RNase A, 在37℃放置30min,以去除RNA。然后加入50μg 蛋白酶K,在37℃放置1h。
(2)加入等体积酚/氯仿/异戊醇(25:24:1),混匀,12000 rpm 离心5 min。
(3)小心吸取上清液到新的1.5 mL离心管,加入 1/10体积的3 mol/L 乙酸钠和2倍体积冰冻无水乙醇,顺时针混匀后,放在-80℃冰箱1h。
(4)12000 rpm 离心 20 min,4℃。
(5)小心去除上清,注意不要吸走DNA沉淀,然后再加入70%乙醇,悬浮沉淀,12000 rpm离心10min,去除上清,让DNA沉淀干燥。
(6)用40 μL TE溶解DNA沉淀。
2.8染色质免疫沉淀DNA的分析和蛋白质在DNA上结合位点的鉴定
(1)如果目的蛋白的靶序列是已知的或者怀疑某个序列是目的蛋白的靶序列,可以根据靶序列设计引物,用荧光定量PCR的方法,比较特异性和非特异性免疫抗体所沉淀的DNA的PCR产物的量,或者比较根据靶序列设计的引物和远离靶序列设计的引物的PCR产物的量。比值越大,则说明靶序列DNA与目的蛋白相结合的可能性越大。
(2)如果目的蛋白的靶序列未知或者高通量研究目的蛋白在基因组的分布情况,找出转录因子的结合位点,可以采用DNA芯片(ChIP-chip)和ChIP-seq方法。
第三部分 目的DNA如何进行检测
3.1实时定量 PCR
用比较精确的实时定量PCR方法检测沉淀的DNA样品,称为实时定量染色质免疫共沉淀技术(qChIP)。这种方法是在体内确定DNA和蛋白质相互作用。与ChIP-on-Chip方法相比,成本低廉。目前实时定量染色质免疫共沉淀技术(qChIP)已经开始应用于分析减数分裂时期蛋白质和DNA相互作用的研究。
3.2染色质免疫共沉淀测序(ChIP-SEQ )
为了在基因组范围内重新发现转录因子的结合位点,需进一步确定染色质免疫共沉淀实验得到的DNA样品的序列。 其序列可以通过直接测序确定,这种方法称为染色质免疫共沉淀测序(ChIP-SEQ)。ChIP-SEQ 为巨大的DNA并行序列应用快速进化平台,以高分辨率确定样品中富集的基因组区域。 目前,Illumina Genome Analyzer(GA)技术频繁用于染色质免疫共沉淀测序(ChIP-SEQ )应用的平台。其它针对高通量测序的平台是 Helicos HeliScope 和 ABI SOL- iD。
3.3 ChIP-on-chip
ChIP-on-chip是将染色质免疫共沉淀(ChIP)和DNA微阵列芯片技术相结合用来高通量分析DNA和蛋白质结合或者翻译后染色质/组蛋白修饰的一种方法。 该技术已经成为深入研究内源蛋白和DNA相互作用的有力工具。因该技术能在基因组范围内确定转录因子或组蛋白修饰所在位置的染色质结合位点,故也被称为全基因组定位分析 (genome-wide location analysis,GWLS)。
该技术是将染色质免疫共沉淀与包含整个基因组或特定目的区域的芯片联合使用,大规模地迅速确定生理条件下特定转录因子在染色体上的结合位点。具体方法是:将经过染色质免疫共沉淀的DNA样品纯化后进行PCR扩增, 然后用荧光素标记(如 Cy3)。对没有经过免疫沉淀反应富集的 Input DNA 样品也进行扩增,并且用另一种荧光素标记(如 Cy5),作为结合背景的参照。之后将这两种 DNA 杂交于包括基因组序列的微阵列芯片上,转录因子结合的靶序列用每个点相应的荧光强度来确定。样品准备是影响ChIP-on- chip 结果的一个重要因素。目前常用的是覆瓦式芯片(tiling arrays),该芯片包含完全非重复性基因组。由于ChIP-on-chip 实验数据量大,问题复杂,所以下游的生物信息学分析相当重要 。 Toedling 等人应用免费和公开的资源 R package Ringo,使 ChIP-on-chip 实验数据的分析更便利。这个软件能提供循环数据输入功能,质量评价,标准化和形象化数据,检测ChIP富集的基因组区域。
ChIP -on -chip 扩展了染色质免疫共沉淀(ChIP)技术的应用范围,可以用于基因组功能的研究,能够绘制基因顺式调控区域和核小体位置及修饰的细节,完善 真核生物的序列。这种方法首次应用在酿酒酵母的研究上,之后被应用于人细胞的研究。在医学方面,已经应用“ChIP-on-chip” 技术确定了骨细胞的动态转录机制。总之,ChIP-on-chip已经成为一种在基因组范围内研究蛋白质和DNA相互作用及转录调控机制的常用方法。 随着各个物种基因组覆瓦式芯片陆续发布,全基因组定位分析技术的应用将会越来越广泛。
3.2-3染色质免疫共沉淀测序(ChIP-SEQ)和ChIP-on-chip技术的比较
ChIP是相对成熟的技术,但目前还存在一些技术难点。例如,ChIP实验涉及的步骤多,结果的重复性较低,需要大量的起始材料;染色质免疫沉淀获得的DNA数量往往很多,包含大量的非特异结合的假阳性结合序列;而对于神经细胞和干细胞等,往往培养困难,并且难以区分个别细胞与总体细胞的表型。在此背景下,配合使用芯片或者第二代高通量测序技术检测这些DNA片段,就形成了ChIP-chip技术和ChIP-Seq技术。
ChIP-Seq是将深度测序技术与ChIP实验相结合分析全基因组范围内DNA结合蛋白结合位点、组蛋白修饰、核小体定位或DNA甲基化的高通量方法,可以应用到任何基因组序列已知的物种,并能确切得到每一个片段的序列信息。相对于ChIP-chip技术,ChIP-Seq是一种无偏向检测技术,能够完整显示ChIP富集DNA所包含的信息。ChIP-chip技术的缺点在于它是一个“封闭系统”,只能检测有限的已知序列信息,相比之下,ChIP-Seq的优势在于其强大的“开放性”,强大的发现和寻找未知信息的能力。因此,ChIP-Seq与传统的ChIP-chip技术相比具有明显的优势:
(1) 灵敏度很高。传统的ChIP-chip实验要求起始DNA的量在4ug以上,而一般ChIP-Seq实验对起始DNA量的要求是10ng。这直接反映在起始细胞数目的减少,对于像早期胚胎发育相关的研究中更占优势。
(2) 灵活性很强。ChIP-chip实验以研究对象的特定物种的全基因组DNA芯片平台为基础,所以不适合应用在那些基因组序列信息不丰富或缺少相关芯片平台开发的物种。ChIP-Seq技术则不存在这方面的限制,可以应用到任何基因组序列已知的物种,并能确切得到每一个片段的序列信息。
(3) 分辨率极高。传统的微阵列芯片技术受制于当前芯片的容量,事实上不能涵盖真正的全基因组DNA序列信息,这导致ChIP-chip的实验结果分辨率不高,精确定位蛋白与DNA的结合位点存在一定的困难。而ChIP-Seq技术辅之以强大的生物信息计算能力,可以高效地将测序得到的序列定位到特定基因组的精确碱基位置上,分辨率大大提升。
(4) 不具备其它一些芯片相关的负效应,如由核酸非特异杂交带来的噪音信号。
因此,随着目前测序的成本不断降低及通量迅速增高等优势,ChIP-Seq已经基本上取代ChIP-chip成为研究转录因子、RNA聚合酶、核小体等DNA结合蛋白体内结合靶点的主打技术。
3.21ChIP-Seq实验设计的关键主要有以下几个方面:
1, 抗体质量:一个灵敏度高和特异性高的抗体可以得到富集的DNA片段,这有利于探测结合位点。
2, 空白对照:空白对照是必要的,存在很多假阳性情况需要通过空白对照进行判断。一般来说有三种类型的空白对照:
(1) 部分进行免疫沉淀前的DNA(input DNA),这是最常用的;
(2) 由免疫共沉淀得到而不含有抗体的DNA(mock IP DNA),使用这个的问题在于收集到的量可能不够;
(3) 使用非特异免疫共沉淀方法得到的DNA。
3, 测序深度:在发表的ChIP-Seq实验中,一般使用Illumina Genome Analyzer上的一个lane产生的数据作为一个基本单位,目前一个lane大概是8-15 million reads。判断足够的测序深度的标准是:当增加测序得到更多的reads时不能发现更多的东西。将这一标准应用到结合位点的数量上就是:进行测序,增加reads数而无法得到更多的结合位点。
4, Multiplexing:对于基因组比较小的物种(E.coli, C.elegans)来说,一个标准的illumina lane得到的数据太多了,仅仅测一个样本比较浪费,所以可以将多个样本加不同的adapter放在一起测。
由于现在提供高通量测序的服务商很多,大家只需要把经ChIP富集得到的DNA样品纯化好交给测序公司就可以了。但是大家经常会遇到测序结果信号弱,背景高等令人头疼的问题,其实ChIP-Seq除了找一家专业的测序服务提供商以外,更重要的是如何获取更高质量的ChIP实验结果。
常见的两种ChIP实验技术有N-ChIP和X-ChIP技术。N-ChIP采用核酸酶消化染色质,适用于研究DNA与高结合力蛋白的相互作用,比如组蛋白修饰等方面的研究;X-ChIP则采用甲醛或紫外线进行DNA和蛋白交联,通过超声波片段化染色质,适合用来研究DNA与低结合力蛋白的相互作用问题,例如大多数非组蛋白方面的蛋白研究。
破碎DNA及具有较高的特异性和亲和力的抗体是ChIP实验成功与否的关键因素。大多数的ChIP实验都使用超声的方法打断DNA,最理想的情况是将DNA打断成200-1000bp的弥散片段,而不同类型及不同数量的细胞对超声的条件都不一样,这往往导致超声的结果无法重复,因此,超声要根据细胞类型和数量对条件进行摸索,将最佳超声条件固定下来,以保持实验的可重复性。
第四部分ChIP实验关键因素有哪些
4.1 甲醛的固定交联程度
甲醛能使蛋白质和DNA固定交联形成复合物,能够真实反映细胞内蛋白质和DNA相互作用情况。最好使用新鲜的甲醛,注意甲醛是否已经过了有效期。刚开始进行染色质免疫沉淀实验时,甲醛固定的时间要进行优化。甲醛固定的时间太长,虽然会得到更多的蛋白质和DNA交联复合物,但是需要更多时间进行超声打碎,使得样品溶液过热导致蛋白质和DNA变性。另外,过度固定交联,会使目的蛋内的抗原决定簇被屏蔽,不能和抗体结合有效结合,而引起免疫沉淀下降。甲醛固定的时间如果太短的话,蛋白质和DNA形成的复合物交联不牢固,很容易在超声时分离,可能在离心步骤时引起丢失。因此要对甲醛的固定时间进行优化,一般的时间为10~20 min。
4.2 超声打碎染色质样本所获得的DNA片段大小
用于染色质免疫沉淀实验的最佳DNA片段要求在超声后为200 ~1000 bp。获得恰当大小的DNA片段关系到实验的成败以及对实验结果的解释。因此要重视超声条件的优化实验。根据不同细胞类型和细胞浓度,选择不同的超声时间和超声次数。最初ChIP实验使用普通的金属探头式超声仪,超声时要把超声针头插入到接近离心管底部的地方,一般离底部5~10 mm,且不要碰到管壁。样本要放在冰上进行操作,以免超声时样品过热而使蛋白和DNA变性。目前大家普遍使用ChIP实验超声神器Bioruptor非接触式超声波破碎仪,由于Bioruptor超声仪一次可以处理多个密闭样品管进行超声处理,样品间不存在交叉污染,超声能量稳定一致,实验重复性大大提高,彻底地完美解决了超声问题。
4.3 抗体能否识别和免疫沉淀目的蛋白的有效性
目的蛋白抗体有效性也是染色质免疫沉淀实验的关键因素。要选择高质量,高亲和力和高效价的抗体来进行实验。先做Western Blot来检验在染色质免疫沉淀实验条件下抗体是否能够和目的蛋白的抗原决定簇结合而免疫沉淀。当抗体不能有效识别交联中的目的蛋白时,可更换其他不同靶向抗原决定簇的抗体或者降低甲醛固定交联时间。
第五部分 超声打断为何推荐要使用Bioruptor超声波破碎仪
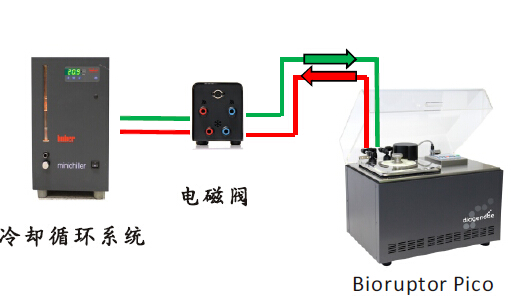
比利时Diagenode生产的Bioruptor plus非接触式细胞破碎仪,其超声源是一块附着在水槽下面的半导体金属块,绝非普通的压电陶瓷。其独特的ACT( Adaptive Cavitation Technology) 聚焦超声技术以及高效的超声效能,能快速处理各种样品,得到满意的超声结果。

仪器标配1.5ml适配器(如上图),可同时处理6个样本,适配器中每个样本的填装体积以及浓度,更是针对ChIP或ChIP-seq技术特别优化过的,与实验室所需的上样量与上样浓度完全衔接,符合实验室需求。
自带电磁阀式冷循环机,与超声仪主机交错启动,可保证在处理测序DNA或chromatin样本片断化时,让样本处于恒定冰冷水浴条件下,同时完全不干扰超声波传到到样本的效率,其高性价比、高通量、高功率、低干扰的优异设计,都是为了让实验的结果更精准、更正确、重复性更佳。
此外更考虑到实验与应用的多元性,厂家特别设计出适合极小量样本的0.65ml适配器,一次可以处理12个样本,是市面上能处理体积最小的适配器,完全是让珍贵样本也能进行ChIP实验特别订制的,也彻底解决了无法处理微量珍贵样品进行ChIP或测序实验的困境。
Diagenode厂家提供了各种实验的方案技巧,可以官网免费下载,或者联系代理商获取,减少因为摸索超声条件而浪费了得之不易的样本。
仪器还含有自动过热断电保护及仪器寿命监控系统,可确保仪器在操作过程中的正常运作以及保护测序样本不过热产生质变的危险,可让仪器正常使用情况可超声效率维持长久不衰减。